- Hardy-Weinberg-Gesetz
-
Das Hardy-Weinberg-Gleichgewicht (HWG) (nach dem Mathematiker G. H. Hardy und dem Arzt Wilhelm Weinberg) ist ein Begriff der Populationsgenetik.
Zur Berechnung dieses mathematischen Modells geht man von einer in der Realität nicht vorzufindenden idealen Population aus, in der sich weder die Häufigkeiten der Allele noch die Häufigkeiten der Genotypen verändern, da diese sich im modellierten Gleichgewicht befinden. Dies bedeutet, dass in einer idealen Population keine Evolution stattfindet, da keine Evolutionsfaktoren greifen und diese den hier konstanten Genpool verändern.
Das Hardy-Weinberg-Gleichgewicht wird trotz seines modellhaften Charakters zum Ableiten von populationsgenetischen Gesichtspunkten vom Modell auf die Realität verwendet. Insbesondere bei sich im Gleichgewicht befindenden Populationen mit relativ großer Größe lässt sich dieses Modell realistisch anwenden. Ferner findet die Regel Anwendung zur Berechnung des Anteils von heterozygoten Individuen (hier im Beispiel: Aa) bei dominant-rezessiven Erbgängen, da heterozygote Organismen von homozygot dominanten (hier: AA) phänotypisch nicht zu unterscheiden sind, da sich das dominante Allel durchsetzt.
Inhaltsverzeichnis
Geschichte
Die Mendelschen Gesetze wurden im Jahre 1900 wiederentdeckt, allerdings wurden sie noch einige Jahre bezweifelt, da man noch keine Aussage fand, wie daraus eine stabile Nachfolgegeneration entstehen kann. Udny Yule argumentierte 1902 gegen die Anwendung[1], da er glaubte, die dominanten Allele müssten sich mit der Zeit in der Population verbreiten. Der US-Amerikaner William E. Castle zeigte 1903 , dass ohne Selektion die genotypischen Häufigkeiten stabil blieben[2]. Karl Pearson, heute bekannt für seine Beiträge zur Statistik, fand 1903 einen Gleichgewichtspunkt bei p = q = 0.5. Der britische Genetiker Reginald Punnett, der Yule's Gegendarstellung nicht widerlegen konnte, befragte seinen Cricket-Spielpartner G. H. Hardy, einen reinen Mathematiker, der die angewandte Mathematik eigentlich verachtete. Im Jahre 1908 veröffentlichte Hardy einen Beitrag[3], in dem er das "sehr einfache" Problem (seine Worte) mit den Begriffen der Biologen erläuterte.
- To the Editor of Science: I am reluctant to intrude in a discussion concerning matters of which I have no expert knowledge, and I should have expected the very simple point which I wish to make to have been familiar to biologists. However, some remarks of Mr. Udny Yule, to which Mr. R. C. Punnett has called my attention, suggest that it may still be worth making...
- Suppose that Aa is a pair of Mendelian characters, A being dominant, and that in any given generation the number of pure dominants (AA), heterozygotes (Aa), and pure recessives (aa) are as p:2q:r. Finally, suppose that the numbers are fairly large, so that mating may be regarded as random, that the sexes are evenly distributed among the three varieties, and that all are equally fertile. A little mathematics of the multiplication-table type is enough to show that in the next generation the numbers will be as (p+q)2:2(p+q)(q+r):(q+r)2, or as p1:2q1:r1, say.
- The interesting question is — in what circumstances will this distribution be the same as that in the generation before? It is easy to see that the condition for this is q2 = pr. And since q12 = p1r1, whatever the values of p, q, and r may be, the distribution will in any case continue unchanged after the second generation
Damit war dieses Prinzip in der englisch-sprachigen Welt als "Hardy's Gesetz" bekannt, bis Curt Stern 1943 darauf verwies[4], dass unabhängig der deutsche Arzt Wilhelm Weinberg es kurz zuvor im Jahre 1908 ebenfalls formulierte[5]. Teils wird auch Castles Name mit hinzugenommen, der das Prinzip früh erkannte, jedoch war seine Formulierung nicht identisch.
Kennzeichen einer idealen Population
- Sehr große Individuenzahl: Der zufällige Verlust eines Individuums oder Gendrift verändert praktisch nicht die Häufigkeit der Allele, was bei einer kleinen Population relativ große Auswirkungen hätte.
- Panmixie: Alle Paarungen, auch von Trägern verschiedener Genotypen, sind gleich wahrscheinlich und gleich erfolgreich.
- Es gibt keine Selektion, somit also weder Selektionsvorteile noch -nachteile für die Träger bestimmter Gene (Genotyp), die sich phänotypisch auswirken.
- Es finden keine Mutationen statt.
- Es finden keine Zu- oder Abwanderungen (Migration) statt, die die Allelfrequenz verändern.
Die ideale Population ist ein theoretisches Konstrukt, da in der Realität mindestens eine der Bedingungen, welche mit Ausnahme der Individuenzahlen alles Evolutionsfaktoren sind, nicht erfüllt wird. Evolution findet also stets dann statt, wenn die obigen Voraussetzungen nicht gelten.
Berechnungsformeln
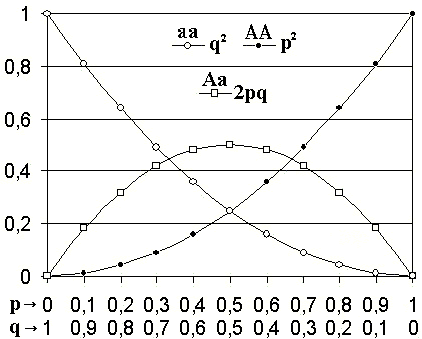
Die beiden Formeln für das Hardy-Weinberg Gleichgewicht lauten:
- p2 + 2pq + q2 = 1
- p + q = 1
Tabelle 1: Punnett-Quadrat für das Hardy–Weinberg-Gleichgewicht Weiblich A(p) a(q) Männlich A(p) AA(p2) Aa(pq) a(q) Aa(pq) aa(q2) - Die Allelfrequenz eines Genpools ist in einer Idealpopulation konstant.
- Die relativen Häufigkeiten der betrachteten Allele sind zueinander komplementär.
- p + q = 1
- p: relative Häufigkeit des Auftretens des Allels A
- q: Allelfrequenz des (zu A komplementären) Allels a
- Genotypfrequenzen (relative Häufigkeit des Auftretens eines Genotyps)
- Die Genotypenfrequenz eines Genpools ist in einer Idealpopulation konstant.
- p2 + 2pq + q2 = 1
- p2 = h(AA)
- 2pq = h(Aa)
- q2 = h(aa)
Mit Hilfe dieser Formeln lässt sich die Häufigkeit eines Allels in einer Population berechnen, wenn die Häufigkeiten der Genotypen bekannt sind bzw. die Häufigkeit eines Genotyps, wenn die Allelfrequenz bekannt ist. Trotz des theoretischen Konstrukts der idealen Population, lassen sich die Formeln durchaus mit Erfolg in der Praxis einsetzen.
Beispiele
Beispiel Blutgruppe
Die Vererbung von Blutgruppen ist kodominant von A und B über 0. Nehmen wir an, die Häufigkeiten der Gene für A, B und 0 im Genpool seien r, s und t (mit r+s+t=1), dann hat ein Anteil von r² den Genotyp AA und ein Anteil von 2rt den Genotyp A0. Die bedingte Wahrscheinlichkeit p (Genotyp=AA unter der Bedingung Blutgruppe=A) ist also r²/(r²+2rt). Diese bedingte Wahrscheinlichkeit ist genau dann gleich 0.5, wie in einer idealen Population, wenn r²=2rt, also dann und nur dann, wenn r=2t. Dieselbe Überlegung für BB bzw. B0 liefert s=2t, also 1=r+s+t=5t, also t=0.2, r=s=0.4. Damit hätte dann ein Anteil von r²=0.16 Genotyp AA, 2rt=0.16 hätte A0, insgesamt also 32% Blutgruppe A. Genauso 32% Blutgruppe B. Für Blutgruppe 0 erhält man t²=0.04, also 4%, für AB 2rs=0.32, also 32%.
Die Werte einer idealen Population im Hardy-Weinberg-Gleichgewicht für den Erbgang kodominanter Gene (A, B, AB zu 32%, 0 zu 4%) tritt bei menschlichen Blutgruppen nicht auf - tatsächlich sind die Werte in den verschiedenen Populationen weltweit sehr ungleich verteilt. Bei Deutschen findet sich 41% "0" und 43% "A" bei nur 11% "B" und 5% "AB", was ähnlich auch in anderen westeuropäischen Völkern zu finden ist. Im Vergleich dazu tritt bei den zentralasiatischen Völkern der Kalmücken und Burjaten die Blutgruppe "B" zu etwa 40% auf, doppelt so hoch wie andere Blutgruppen. Weltweit ist "0" die häufigste Blutgruppe, die z.B. bei südamerikanischen Indianern bis zu 100% ausmacht.
Die Kenntnis des Erbganges und des Wertes einer idealen Population im Hardy-Weinberg-Gleichgewicht gibt einen Hinweis, dass bestimmte Gene in einem Genpool einem Selektionsdruck oder Migrationseinflüssen ausgesetzt waren - am stärksten für das rezessive Gen "0", dem man einen Schutz vor Syphilis zuschreibt.
Beispiel Phenylketonurie
Die Häufigkeit für das Auftreten der Erbkrankheit Phenylketonurie in der Bevölkerung beträgt 1:10000. Alle Kranken besitzen zwei (rezessive) Allele a; wer mindestens ein (dominantes) Allel A besitzt, erkrankt nicht. Die oben angegebene Häufigkeit ist somit identisch mit der Frequenz des Genotyps 'aa': h(aa) = 0,0001
Damit ist q = 0,01 (als Wurzel aus h(aa)) und p = 0,99 (wegen p + q = 1). D.h. auch: 1% aller Gene (zu den Allelen A und a) in der Bevölkerung sind 'defekt'.
Alle Träger des (heterozygoten) Genotyps 'Aa' sind Überträger des defekten Allels a. Ihre Häufigkeit in der Bevölkerung beträgt h(Aa) = 2 pq = 0,0198 - das sind annähernd 2%.
Von 10000 Individuen haben also (theoretisch)
- 9801 den Genotyp AA und sind phänotypisch gesund;
- 198 den Genotyp Aa und sind zwar phänotypisch gesund, können aber das krankmachende Allel an die nächste Generation weitergeben;
- 1 den Genotyp aa und ist an Phenylketonurie erkrankt.
q²= 1:10000, damit q=1:100 Heterozygotenhäufigkeit: 2x1(=0,99)x0,01 = 0,02
Beispiel Rot-Grün-Sehschwäche
Die Rot-Grün-Sehschwäche wird X-chromosomal rezessiv vererbt. Die Krankheit tritt dann auf, wenn ein defektes X-Chromosom vorliegt, welches nicht durch ein intaktes X-Chromosom überdeckt wird. Es seien 9 % der Männer von der Rot-Grün-Sehschwäche betroffen. Wie viele Frauen sind dann betroffen?
- Lösung
Es gibt 2 Allele für das X-Chromosom:
- X = keine Sehschwäche
- x = defektes X-Chromosom → Sehschwäche
- X ist dominant gegenüber x
- 91 % aller X-Chromosomen bei Männern haben das Allel X
- 9 % aller X-Chromosomen bei Männern haben das Allel x
Bezogen auf beide Geschlechts-Chromosomen ergibt das folgende Häufigkeit bei Männern:
- p = h(Y) = 0,5
- q1 = h(X) = 0,5 * 0,91 = 0,455
- q2 = h(x) = 0,5 * 0,09 = 0,045
Mögliche Genotypen der Männer: XY und xY Häufigkeit innerhalb der Population: 0,91 und 0,09 Mögliche Keimzellen: X Y bzw. x Y Häufigkeiten dieser Keimzellen: 0,5*0,91 0,5*0,91 0,5*0,09 0,5*0,09 - Häufigkeit des defekten x-Chromosoms im Frauengenpool = q = h(x) der Frauen
- Bei einer idealen Population werden in der nächsten Generation wieder 9 % kranke männliche Nachkommen geboren, das sind 4,5 % bezogen auf alle Kinder. Da männliche Nachkommen das x- oder X–Chromosom nur von der Mutter erhalten, ist q = 9%.
Zusammenfassung Genotyp:
Frauen X (p = 0,91) x (q = 0,09) Männer Y (p1 = 0,5) XY (p * p1 = 0,455) xY (q * p1 = 0,045) X (q1 = 0,455) XX (p * q1 = 0,41405) xX (q * q1 = 0,04095) x (q2 = 0,045) Xx (p * q2 = 0,04095) xx (q * q2 = 0,00405) Die Rot-Grün-Sehschwäche tritt bei Frauen phänotypisch nur beim Auftreten von 2 defekten x-Chromosomen auf. Das sind 0,405 % bezogen auf alle Kinder oder 0,81 % bezogen auf die weiblichen Nachkommen.
Siehe auch
Weblinks
Quellen
- ↑ G.U. Yule : "Mendel's laws and their probable relation to intra-racial heredity", New Phytol. 1, 1902
- ↑ W. E. Castle: "The laws of Galton and Mendel and some laws governing race improvement by selection", Proc. Amer. Acad. Arts Sci.. 35, 1903
- ↑ G. H. Hardy: "Mendelian proportions in a mixed population". Science 28, 1908
- ↑ C. Stern: "The Hardy–Weinberg law". Science 97, 1943
- ↑ Wilhelm Weinberg: "Über den Nachweis der Vererbung beim Menschen", Jahrhefte des Vereines für Vater. Naturkunde in Württemberg, 1908
Wikimedia Foundation.