- DNA-Reparaturmechanismus
-
Durch DNA-Reparatur-Mechanismen können Zellen schadhafte Veränderungen der DNA-Struktur in einer Zelle beseitigen. Solche Schäden in der DNA können spontan im Verlauf der DNA-Replikation oder durch die Einwirkung mutagener Substanzen, extremer Wärme oder ionisierender Strahlung verursacht werden.
DNA-Schäden können dazu führen, dass die Replikation der DNA für die Mitose falsch erfolgt, Proteine nicht mehr bzw. falsch synthetisiert oder wichtige Chromosomenbereiche nach Doppelstrangbrüchen abgespalten werden.
Bringen die komplexen Reparaturmechanismen der Zelle keinen Erfolg, so sammeln sich in wachsenden und ruhenden somatischen Zellen so viele Fehler an, dass die normalen Zellfunktionen gestört sind. In einer Keimzelle wären die Tochterzellen nicht mehr lebensfähig, was zu einer Inaktivierung der Zelllinie führt: die Zelle bzw. die zweite bis dritte nachfolgende Generation verliert ihre Teilungsfähigkeit und stirbt. Im Zuge der Zellzykluskontrolle können Kontrollproteine eine Zelle bzw. deren DNA als defekt erkennen und einen Zyklusarrest oder den programmierten Zelltod (Apoptose) einleiten.
Inhaltsverzeichnis
Ursachen von DNA-Schäden
Mögliche Ursachen sind Stoffwechselvorgänge, UV-Strahlung und Ionisierende Strahlung.
Stoffwechselvorgänge
Eine Zelle ist ein System im Fließgleichgewicht. Sie nimmt fortwährend Moleküle auf, verarbeitet sie, synthetisiert benötigte Stoffe, und gibt wiederum bestimmte Stoffe an die Umgebung ab. Beim normalen zellulären Metabolismus können reaktive Sauerstoffspezien (ROS, unter anderem Sauerstoffradikale) entstehen, welche ein signifikantes Ausmaß an oxidativen Schaden anrichten. Am häufigsten sind dies Basenschäden und Einzelstrangbrüche, weniger als 0,5 % sind Doppelstrangbrüche, welche auch noch relativ gleichförmig über die DNA verteilt sind. Die Wahrscheinlichkeit endogen induzierter Schadenscluster und damit – schwierig zu reparierender – gehäufter Läsionen (complex lesions), wie sie sonst durch die nicht-homogene Energieabgabe ionisierender Strahlen auftreten, ist sehr gering.
UV-Strahlung
Durch die UV-Strahlung kann es zu direkten Veränderungen (Mutationen) der DNA kommen, wobei diese insbesondere UV-B-Strahlung absorbiert. Sowohl UV-B als auch UV-A können indirekt die DNA durch die Entstehung von reaktiven Sauerstoffradikalen schädigen, die die Entstehung von Oxidativen DNA-Läsionen bewirken, die wiederum zu Mutationen führen. Diese sind vermutlich für die Entstehung von UV-A-induzierten Tumoren verantwortlich.[1]
Arten von DNA-Schäden
- Pyrimidindimere zu 75%
- 6-4-Photoprodukt zu 21%
- Basenschaden zu 65% nach Röntgenbestrahlung
- Einzelstrangbruch zu 25%
- Gehäufte Läsionen zu 4%
- DNA-Protein-Vernetzung zu 4%
- Doppelstrangbruch zu <1%
Reparatur von DNA-Schäden
Basenexzisionsreparatur
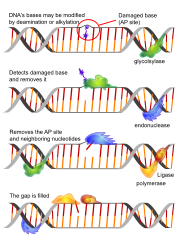
Bei der Basenexzisionsreparatur wird ein Fehler in der Basenpaarung eines der beiden DNA-Stränge behoben. Der Fehler kann von Reparaturenzymen erkannt werden, weil die Spiral-Struktur der DNA an der fehlerhaften Stelle von der Norm abweicht. Die betreffende Base wird von einer DNA-Glykosylase entfernt, danach wird durch eine AP-Endonuklease (A-Purin-A-Pyrimidin-Endonuklease) die nun nicht mehr besetzte Desoxyribose ausgeschnitten. Eine DNA-Polymerase synthetisiert abhängig von der komplementären Base auf dem fehlerfreien Strang die korrekte Base. Eine DNA-Ligase verankert die neue Base im DNA-Strang, womit der Fehler korrigiert ist.
Nukleotidexzisionsreparatur
Es gibt zwei verschiedene Formen der Nukleotidexzisionsreparatur (NER). Zum einen Global Genome Repair (GGR), welche Schäden in transkriptionsinaktiven Bereichen der DNA behebt und zum anderen die sogenannte Transcription Coupled Repair (TCR), welche Schäden an der aktuell zu transkribierenden DNA behebt. Diese beiden Formen unterscheiden sich nur in der Schadenserkennung. Bei der GGR wird die DNA-Läsion vom Proteinkomplex XPC/HHR23B erkannt. Dagegen spielt dieser Komplex bei der TCR keine Rolle. Bei der TCR ist es von Bedeutung, dass die durch die Schädigung blockierte RNA Polymerase II entfernt wird, um so den TCR-Proteinen Zugriff zur DNA-Schädigung zu ermöglichen. Dieses Entfernen der RNA Polymerase II wird durch CSA und CSB ermöglicht. Die weiteren Schritte sind bei beiden Reparaturwegen identisch. XPA und RPA dienen zur weiteren DNA-Schadenserkennung und dirigieren die Helikasen XPB und XPD zur Läsion, welche die DNA unmittelbar in der Nähe der Schädigung entwinden. Die Endonukleasen XPG und XPF/ERCC1 schneiden den DNA-Strang mit der Schädigung in 3´ und 5´ Richtung, so dass ein 30 Basen umfassendes Oligonukleotid freigesetzt wird, welches die Schädigung enthält. Nun folgt die Polymerisation des fehlenden DNA-Abschnitts durch die DNA Polymerase β und weitere Faktoren. Als letztes erfolgt die Ligation des synthetisierten Abschnitts durch die DNA-Ligase I. Mutationen, welche die CSA/B Gene betreffen, führen zur Ausbildung des Krankheitsbildes Cockayne-Syndrom. Mutationen betreffend der XPA-XPG Familie führen zur Ausbildung des Krankheitsbildes Xeroderma pigmentosum.
Korrekturlesen durch DNA-Polymerase (Basenfehlpaarungsreparatur, Mismatch-Reparatur)
Das für das Kopieren der DNA zuständige Protein DNA-Polymerase besitzt die Fähigkeit, den neuen DNA-Strang noch während der Synthese zu überprüfen und mit dem ursprünglichen Strang zu vergleichen. Entdeckt es fehlerhafte Nukleotide, werden diese herausgeschnitten und neu synthetisiert. In Eukaryonten übernehmen die δ- und die ε-DNA-Polymerasen diese Aufgabe, in Prokaryonten die Polymerase III (3'-5'-Exonukleaseaktivität). Ist die Polymerase mutiert und verliert dadurch die Korrekturlesefunktion, steigt die Anzahl spontaner Mutationen um das 1000-fache. Das Bakterium Escherichia coli kann zudem anhand des Methylierungsstatus den bei der Replikation entstandenen fehlerhaften Tochterstrang von der abgeschriebenen DNA unterscheiden. Der neue Strang wird etwas später als die Matrize (der Elternstrang) an den Adeninresten der Sequenz GATC methyliert. Ein Defekt in der Mismatchreparatur verursacht eine Form von Darmkrebs: HNPCC (Hereditäres-Nichtpolypöses-Colon-Carzinom).
Photoreaktivierung
Photolyasen sind in der Lage, durch ultraviolette Strahlung in der DNA entstandene Cyclobutan-Ringe und (6-4)-Photoprodukte aufzulösen. Sie verfügen über einen sog. Antennenkomplex, mit dem sie blaues oder ultraviolettes Licht absorbieren und mithilfe dieser Energie vom Kofaktor FAD zwei Elektronen auf den im aktiven Zentrum gebundenen DNA-Schaden übertragen. Selbiger spaltet sich in der Folge. Das Enzym stellt so ohne Herausschneiden und Einfügen von Basen die native Struktur der DNA wieder her.
Reparatur von Doppelstrangbrüchen (Rekombinationsreparatur)
Bei einem Doppelstrangbruch gibt es die Möglichkeit, dass das Schwesterchromosom die fehlende Information für diesen DNA-Abschnitt überträgt.
- Nichthomologes Endjoining
- Homologe Rekombination
- Single strand annealing
Eine Störung dieses Reparatursystems führt häufig zum Nijmegen-Breakage-Syndrom.
Einzelnachweise
- ↑ P. Elsner et al: Täglicher Lichtschutz in der Prävention chronischer UV-Schäden der Haut. Leitlinie der Deutschen Gesellschaft für Dermatologie, 2005. (PDF, 260 KB)
Weblinks
Wikimedia Foundation.