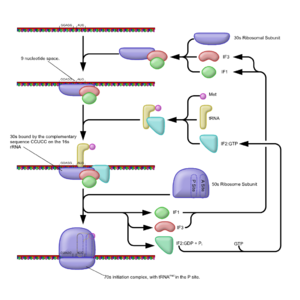- Initiationsfaktoren
-
Initiationsfaktoren (genauer: Translations-Initiationsfaktoren) sind Proteine und Proteinkomplexe in den Zellen aller Lebewesen, die notwendig für eine effiziente Translation der mRNA sind und eine Funktion während der Initiation der Translation haben. Da sich die Initiation der Eukaryoten wesentlich von der der Prokaryoten unterscheidet, müssen die daran teilnehmenden Proteine differenziert werden. Mutationen in den Genen, die für Untereinheiten des eIF-2B-Komplexes codieren, sind für eine spezielle erbliche Form der Leukodystrophie (VWM) verantwortlich.[1]
Inhaltsverzeichnis
Prokaryotische Initiationsfaktoren
Den Prokaryoten genügen drei Proteine, um die Translation einzuleiten. Der Initiationsfaktor IF-1 verhindert zunächst das vorzeitige Binden der tRNA und der Ribosom-Untereinheiten, bis IF-2 zusammen mit Formylmethionin-mRNA andockt. IF-3 ist optional und kann verschiedene Funktionen haben.
Eukaryotische Initiationsfaktoren
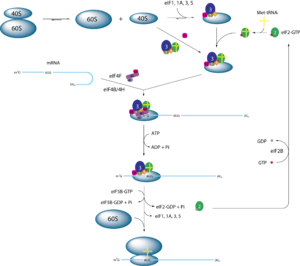
Eukaryotische Initiationsfaktoren gehören zu vielen verschiedenen Proteinfamilien und haben eine Vielzahl von Funktionen: die mRNA zu entwinden, ihre 5'-Cap-Struktur zu erkennen, ihre Bindung an das Ribosom zu regulieren, die Starter-tRNA zu binden und den Komplex zu verlassen, damit die Ribosom-60S-Untereinheit die Elongation durchführen kann.[2]
Beim Menschen sind etwa 45 Gene bekannt, die für eukaryotische Initiationsfaktoren codieren. Die Proteine gehören zu den folgenden Komplexen und Familien:[3]
Komplex Protein-
familieGene (HGNC) Funktion DEAD-Box-Helikasen, DEAH-Familie DHX29 Bindet an 40S nahe des mRNA-Eingangs; Effizienz der Translation eIF-1A EIF1AX, EIF1AY Hilft bei der Trennung der Ribosom-Untereinheiten, bindet Met-tRNA; Effizienz der Translation eIF-2 eIF-2α EIF2S1 Komplex mit GTP und Initiator-tRNA, später zusätzlich mit 40S, verbraucht GTP eIF-2β EIF2S2, A6NK07, EIF5 eIF-2G EIF2S3, EIF2S3L eIF-2B eIF-2B-α/β/δ EIF2B1, EIF2B2, EIF2B4 Komplex katalysiert GTP-GDP-Austausch in eIF-2. Pathologie: Leukodystrophie eIF-2B-γ/ε EIF2B3, EIF2B5 eIF-2A EIF2A Ermöglicht Initiation bestimmter spezieller mRNA eIF-2D EIF2D Verschafft tRNA zur P-Site der 40S, ohne GTP zu benötigen. Außerdem Elongationsfaktor. eIF-3 div. Modul A: EIF3A, EIF3B, EIF3G, EIF3I. Modul B: EIF3F, EIF3H, EIF3M. Modul C: EIF3C, EIF3D, EIF3E, EIF3L, EIF3K Bindet an 40S, ermöglicht Bindung von eIF-1/2, stimuliert Initiator-tRNA-Bindung und 5'-Cap-Erkennung; erleichtert schlussendlich Auftrennung des Gesamtkomplexes, damit 60S andocken kann. eIF-4F DEAD-Box-Helikasen, eIF4A-Familie EIF4A1, EIF4A2 Erkennt Cap-Struktur, ermöglicht mRNA-Bindung an Ribosom, entwindet mRNA. eIF-4E EIF4E, EIF4E1B, EIF4E2, EIF4E3 eIF-4G EIF4G1, EIF4G2, EIF4G3 eIF-6 EIF6 Bindet an 60S und verhindert deren Andocken an 40S IF-2 MTIF2, EIF5B Initiationsfaktor in Mitochondrien; homolog zu prokaryotischen IF-2; erleichtert Bindung von Formylmethionin-tRNA IF-3 MTIF3 Initiationsfaktor in Mitochondrien; homolog zu prokaryotischen IF-3; erleichtert Dissoziation der 55S-Ribosomen Literatur
- T. V. Pestova, V. G. Kolupaeva u.a.: Molecular mechanisms of translation initiation in eukaryotes. In: Proceedings of the National Academy of Sciences of the United States of America. Band 98, Nummer 13, Juni 2001, S. 7029–7036. doi:10.1073/pnas.111145798. PMID 11416183. PMC 34618. (Review).
- K. Asano, L. Phan u.a.: A multifactor complex of eIF1, eIF2, eIF3, eIF5, and tRNA(i)Met promotes initiation complex assembly and couples GTP hydrolysis to AUG recognition. In: Cold Spring Harbor symposia on quantitative biology. Band 66, 2001, S. 403–415, ISSN 0091-7451. PMID 12762043. (Review).
- A. G. Hinnebusch: eIF3: a versatile scaffold for translation initiation complexes. In: Trends in biochemical sciences. Band 31, Nummer 10, Oktober 2006, S. 553–562, ISSN 0968-0004. doi:10.1016/j.tibs.2006.08.005. PMID 16920360. (Review).
- F. Saletta, Y. S. Rahmanto, D. R. Richardson: The translational regulator eIF3a: the tricky eIF3 subunit! In: Biochimica et biophysica acta. Band 1806, Nummer 2, Dezember 2010, S. 275–286. doi:10.1016/j.bbcan.2010.07.005. PMID 20647036. (Review).
Einzelnachweise
- ↑ Leukoencephalopathy with vanishing white matter bei Online Mendelian Inheritance in Man
- ↑ Jochen Graw, Wolfgang Hennig: Genetik 2010, 5. Aufl., Springer 2010. ISBN 3642049982. S.84-85
- ↑ UniProt Suchergebnis Initiationsfaktoren Mensch
Kategorien:- Proteingruppe
- Proteinkomplex
- Molekularbiologie
- Genetik
- Genexpression
- Krankheitsassoziiertes Protein
Wikimedia Foundation.